Introduction
Les acides nucléiques sont des macromolécules présentes dans tout le règne vivant et également les virus. Les acides nucléiques sont des polymères de nucléotides : en fonction du type de nucléotide, on distingue l’acide désoxyribonucléique ($\mathrm{ADN}$) et les acides ribonucléiques ($\mathrm{ARNs}$).
Les acides nucléiques sont soit le support de l’information génétique ($\mathrm{ADN}$), soit participent à son expression sous forme de protéine, ou à sa régulation ($\mathrm{ARNs}$).
Les nucléotides ont un rôle essentiel dans l’activité métabolique de la cellule : cofacteurs, transfert d’énergie, phosphorylation des protéines, régulation de canaux ioniques, …
Les bases azotées
Schéma général des nucléotides (voir figure).
Il existe $5$ bases impliquées dans les acides nucléiques. Elles sont réparties en deux familles (voir figure) :
- Les purines : adénine et guanine ;
- Les pyrimidines : cytosine, thymine (uniquement $\mathrm{ADN}$), uracile (uniquement $\mathrm{ARN}$).
Le sucre associé aux bases est le ribose ($\mathrm{ARNs}$) ou le désoxyribose ($\mathrm{ADN}$) (voir figure). Les fonctions chimiques du sucre, associées au carbone $3’$ (fonction alcool) et au carbone $5’$ (acide phosphorique) joueront un rôle essentiel dans la polymérisation des nucléotides pour construire les acides nucléiques.
$1$ à $3$ acides phosphoriques peuvent être associés à un nucléoside (base + sucre) pour former un nucléotide : $1$ acide phosphorique = nucléotide mono-phosphate (Ex. AMP) ; $2$ ac. phos. = nu. diphosphate (état transitoire) ; $3$ ac. phos. = nucléotide triphosphate ($\mathrm{NTP}$) (Ex. $\mathrm{ATP}$). Lors de leur intégration dans un acide nucléique, les nucléotides sont toujours sous forme $\mathrm{NTP}$.
L’ADN
Polymère de désoxyribonucléotides. Il est formé de deux brins positionnés de façon antiparallèle. Les polymères vont de quelques milliers de paires de bases (pb) (bactéries, virus) à $10^{11}$ pb chez les plantes.
Liaison intrabrin : liaison phosphodiester entre la fonction alcool en $3’$ du nucléotide $n$, avec l’acide phosphorique en $5’$ du nucléotide $n+1$. L’enchaînement entre les nucléotides est libre : c’est la séquence d’$\mathrm{ADN}$. Chaque individu, chaque espèce possède sa propre séquence d’$\mathrm{ADN}$ même si des sections sont conservées. Seuls les jumeaux homozygotes et les clones (bactérie, virus et plante) ont théoriquement exactement la même séquence en bases.
Liaison interbrin : l’$\mathrm{ADN}$ existe uniquement sous forme double-brin, dans les conditions naturelles. Des liaisons hydrogène entre les bases des deux brins permettent d’associer les deux brins. Seulement, tous les couples de bases ne sont pas possibles : $\mathrm{A}$ associé forcément à $\mathrm{T}$ et $\mathrm{G}$ associé forcément à $\mathrm{C}$. Les deux brins sont dits complémentaires.
Les brins ont une orientation, qui dépend des fonctions libres des nucléotides placés aux extrémités : fonction $3’$ ou $5’$. On indique à chaque extremité du brin, le caractère $5'$ ou $3'$ approprié.
La molécule d’$\mathrm{ADN}$ n’est pas linéaire dans la nature : c’est une double-hélice (deux brins enroulés). Les enroulements des deux brins provoquent deux « creux » : grand sillon et petit sillon. Ils sont essentiels pour les interactions avec les protéines. Le modèle de double-hélice le plus courant est le modèle $\mathrm{B}$ : hélice droite, $\mathrm{1.2~nm}$ de diamètre, $10$ nucléotides par tour, $\mathrm{0.34~nm}$ entre chaque nucléotide. Il existe également les formes $\mathrm{A}$ et la $\mathrm{Z}$.
L’$\mathrm{ADN}$ se trouve dans le noyau de la cellule eucaryote : sous forme décondensé (euchromatine) ou sous forme condensée à différents degrés (hétérochromatine). La forme ultime de condensation de l’$\mathrm{ADN}$ est le chromosome (uniquement quand la cellule se divise). ATTENTION : le noyau humain contient $46$ molécules d’$\mathrm{ADN}$ chacune correspondant à un chromosome bien particulier.
Chez les bactéries (Procaryotes), l’$\mathrm{ADN}$ se trouve dans le cytoplasme, sous la forme d’une molécule circulaire = chromosome bactérien. Il peut être associé à d’autres molécules d’$\mathrm{ADN}$ circulaire mais qui contiennent uniquement une information génétique complémentaire (résistance aux métaux lourds, aux antibiotiques, possibilité de conjugaison) : ce sont des plasmides.
Les ARNs
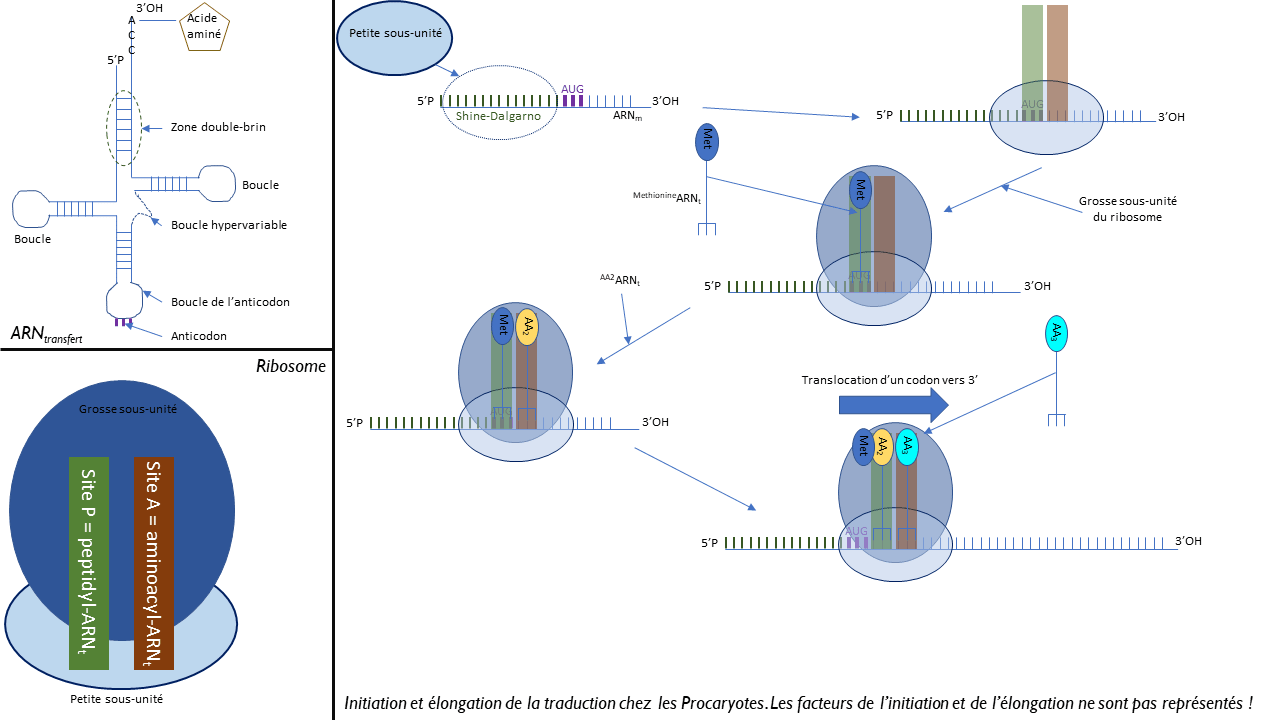
Polymères de ribonucléotides. Ils sont simples brins, dans la Nature, mais peuvent présenter des zones doubles-brins, par repliement du brin sur lui-même, au niveau de séquences complémentaires ($\mathrm{ARN_{transfert}}$ et $\mathrm{ARN_{ribosomique}}$). Les $\mathrm{ARN_{messager}}$ ne présentent pas de repliement.
Dans les $\mathrm{ARNs}$, le nucléotide $\mathrm{T}$ est remplacé par le nucléotide $\mathrm{U}$, sans que la complémentarité avec $\mathrm{A}$ ne soit affectée.
Les $\mathrm{ARNs}$ sont beaucoup plus courts que l’$\mathrm{ADN}$ et leur stabilité est réduite, en raison de la structure simple brin.