La traduction est l’ultime étape de l’expression du génome et consiste en la production d’une protéine à partir d’une matrice constituée d’ARNm. Elle se déroule dans le cytoplasme des Procaryotes, dans le cytoplasme ou le réticulum endoplasmique rugueux pour les Eucaryotes.
Le code génétique
Le mot « traduction » est à prendre au sens littéral : on transforme un langage en un autre langage. Concrètement, on passe d’un message en nucléotides (la séquence de l’ARNm) à un message en acides aminés (la séquence de la protéine). Il existe donc un code qui fait correspondre à chaque nucléotide le bon acide aminé.
Il faut pouvoir coder les 20 acides aminés différents, donc il faut une combinaison de nucléotides qui permet d’obtenir au moins 20 solutions. Si on considère un nucléotide, on ne peut coder que 4 solutions. Si on considère 2 nucléotides, on ne peut coder que 42 solutions, soit 16 acides aminés. Si on prend 3 nucléotides (triplet), on peut coder 43, soit 64 solutions, ce qui est supérieur au nombre d’acides aminés. Chaque triplet est appelé codon. La Nature ayant horreur du vide, et pour éviter les ambiguïtés, les acides aminés sont codés par plusieurs triplets différents. Aucun triplet n’a pas de code associé. Il existe également trois triplets spéciaux qui sont des non-sens = codon stop : ils signifient au ribosome que la traduction s’arrête et que la protéine est terminée.
Le code génétique est universel : tous les organismes (des virus à H.sapiens) utilisent le même.
Les ARNtransfert
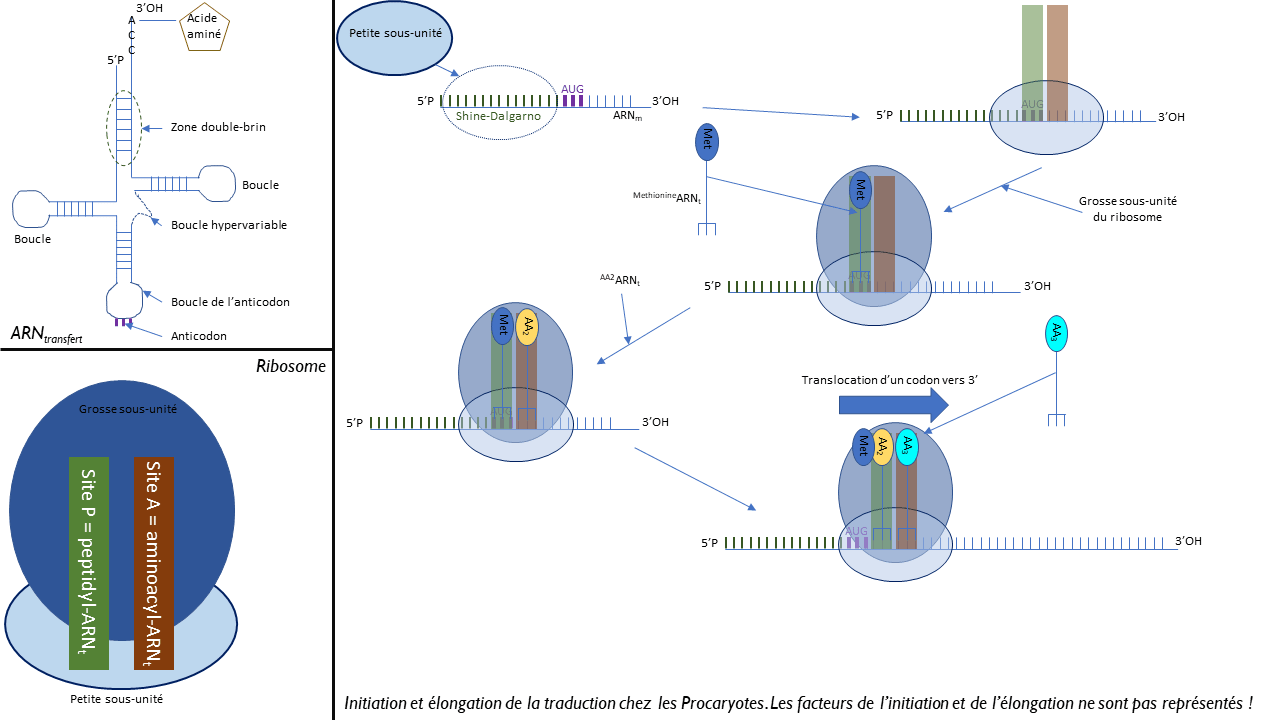
Les ARNtransfert (ARNt) sont les molécules qui font le lien entre les deux langages (nucléotides et acides aminés). Ils possèdent deux sites majeurs (voir figure) :
- Un site de fixation de l’acide aminé, sur l'extrémité 3′−OH ;
- Une boucle anti-codon (complémentaire du codon correspondant à l’acide aminé).
Lors de la lecture de la séquence en nucléotides de l’ARNm, la machinerie cellulaire (ribosome) fait correspondre le codon lu et l’anticodon porté par l’ARNt. Ainsi, l’acide aminé mis en place respectera le sens de l’information génétique.
La synthèse des ARNt est extrêmement complexe et mobilise les aminoacyl-ARNt-synthétases.
Les ribosomes
Complexes nucléoprotéiques, constitués de 65% d’ARNribosomaux (ARNr) et de 35% de protéines. Ils sont définis en fonction de leur constante de sédimentation : grandeur en svedberg (S). Les complexes sont dissociables en deux particules :
- Procaryotes : ribosome 70 S, dissocié en particules 50 S et 30 S ;
- Eucaryotes : ribosome 80 S, dissocié en particules 60 S et 40 S.
Les ARNr sont généralement très conservés et permettent de réaliser des analyses de phylogénie entre les espèces : on parle souvent des ARNr 16 S chez les Procaryotes (il en existe d’autres).
Le ribosome possède deux chambres (voir figure) : site A et site P. Le site A est le site qui accueille le nouvel ARNt associé à son acide aminé. Le site P contient le dernier ARNt dont l’acide aminé a été fixé au peptide en production. Le peptide est d’ailleurs fixé sur l’acide aminé de cet ARNt.
Déroulement de la traduction chez les Procaryotes
Chez les Procaryotes, un ARNm est transcrit et traduit en même temps, par plusieurs ribosomes.
Initiation : reconnaissance par la petite sous-unité du ribosome (30 S) d’une séquence signal située en amont du codon initiateur = séquence Shine-Dalgarno (voir figure). La petite sous-unité est fixée de telle sorte que :
- Le site P soit placé sur le codon initiateur de la traduction ;
- Le site A soit placé sur le codon suivant.
Le codon initiateur est toujours le codon AUG, il correspond à la méthionine : une méthionine modifiée (formyl-méthionine) se fixe en acide aminé 1 (AA1). Une fois l’ARNt de la méthionine fixé dans le site P, la grosse sous-unité (50 S) se fixe à son tour sur la petite sous-unité : le ribosome est complet.
Ces étapes nécessitent des facteurs d’initiation (Initiation Factors) et de l’énergie sous forme de GTP.
Elongation : succession de trois étapes qui se répètent jusqu’à l’arrivé du site A au codon « STOP » ;
- Fixation de l’ARNt correspondant au codon faisant face au site A ;
- Fixation de l’acide aminé (ou peptide en formation) de l’ARNt du site P sur l’acide aminé de l’ARNt du site A. Cette réaction est catalysée par la peptidyl-transférase, située dans 50 S (on déplace bien un peptide !). L’enzyme forme une liaison peptidique ;
- Translocation du ribosome de 3 nucléotides vers 3′(=1 codon), avec éjection de l’ARNt du site P, remplacé par l’ARNt du site A. Le site A se retrouve vide. Retour à l’étape 1.
Ces étapes consomment de l’énergie, deux GTP par cycle, et mobilisent des facteurs protéiques de l’élongation : Elongation Factors.
Terminaison : lorsque le site A se trouve en face du codon « STOP », aucun ARNt ne peut se fixer dans le site A. Cette situation, reconnue par des facteurs de terminaison (Release Factors) provoque la libération de la protéine et le décrochage du ribosome.
La méthionine initiale peut être excisée si la séquence finale de la protéine ne comporte pas de méthionine en position 1.
Déroulement de la traduction chez les Eucaryotes
Initiation : il n’existe pas de séquence Shine-Dalgarno chez les Eucaryotes. La tête de , fixée en de l’, fait office de rail pour la petite sous-unité du ribosome.
Elongation : elle se déroule soit dans le cytoplasme, soit dans le réticulum endoplasmique (). Dans ce cas (), la séquence de la protéine présente, dès le début de sa formation, une succession d’acides aminés reconnus par la (particule de reconnaissance du signal). se fixe sur la séquence, et « force » le ribosome à se rapprocher du , en se fixant sur un récepteur à . A ce moment-là, le peptide produit est libéré, au fur et à mesure de sa formation, dans la lumière du , grâce à un pore. Les protéines produites dans le sont destinées au , Golgi, lysosomes ou voie de sécrétion.
Les étapes d'élongation et de terminaison sont identiques à celles observées chez les Procaryotes.
Adressage : pour les protéines produites dans le cytoplasme, des séquences signal permettent de les diriger vers leur compartiment de travail (noyau, mitochondries, chloroplastes ou peroxysomes).
Modifications post-traductionnelles : excision de la méthionine initiale, ajout de glycosylations sur les protéines, mise en place de la structure tertiaire assistée par des protéines chaperonnes.